bwa によるマッピング
- 概要: bwa とは
- bwa のインストール
- bwa の使い方
- インデックスの作成
- マッピング
- BWA のパラメーター
広告
概要: bwa とは
bwa は、bowtie2 などと似たマッピングプログラムである。Burrow Wheeler Aligner の略であるはずだが、BWA alighner という表現も見かける。BWA には、以下のような特徴がある。
- リファレンス配列を Burrows-Wheeler 変換しているため、高速かつメモリ消費が低い (2)。
- デフォルトで bowtie2 よりもミスマッチの許容度が大きい。たとえば aln を実行すると、225bp reads: max_diff = 9 などの設定が最初に出力される。bowtie2 は 0 か 1 しか選べない。
- イントロンのような長い挿入があると、マッピングに失敗する (2)。そのため、真核生物の RNA-seq データにはほとんど使われておらず、ゲノムリードのマッピングによく使われる。
bwa のインストール
Ubuntu 20.4 では sudo apt install bwa で 大丈夫だった。GitHub も参照のこと。
Mac では、上記の github からフォルダをダウンロードし、make でコンパイルする。リンク先を参照のこと。
広告
「あとがき」で当サイトを参考にしたと書いてくれているラノベです。Kindle Unlimited で読めました。ストーリーと文章が良く、面白かったです。
bwa の使い方
インデックスの作成
-a でアルゴリズムを is または bwtsw から選択。is は 2G 以下の小さなゲノム用。細菌などのデータ解析、bwtsw よりも高速。
それ以上の大きなゲノムには bwtsw を使う。
インデックスの名前を指定したい場合、-p オプションを使う。-p indexname として指定。
マッピング
BWA には 3 つのマッピングモードがある。
backtrack |
bwa aln で実行する。100 bp 程度までのショートリードのマッピングに使う (2)。 |
bwasw |
ロングリードのマッピング。70 から数 Mb までマッピング可能。 |
mem |
同じく 70 から数 Mb までのロングリードのマッピングで、ギャップが多いと予想される場合。 |
Single-end の場合は、以下のようにして実行する。aln/bwasw/mem から 1 つを選択。
Paired-end の場合は、単に 2 つのリードが含まれるファイルを並べる。
アウトプットは bam または sam ファイルになる。一度、間違えて > output.bam の形で出力したところ、ファイルは生成されたが、どうやっても情報を取り出すことができなかった。samtools の使い方 のページも参照のこと。
BWA のパラメーター
mem, bwasw など、どのプログラムを使うかによってパラメーターが変わる。詳細は マニュアル を参照のこと。 [3] のように [ ] の中に書かれている値がデフォルトである。
私がいじったことがあるパラメーターのみを表にしておく。bwasw のパラメーターである。
a |
-a INT で score of match つまりマッチしたときに加算されるスコアを指定する。デフォルトは 1。 |
b |
-b INT で mismatch penalty を指定する。デフォルトは 3。 |
q |
-q INT で gap open penalty を指定する。デフォルトは 5。 |
これらのパラメーターをいろいろ変えて、マッピングがどうなるか試してみたことがある。
a = 2, b = 1, q = 1。とにかくマッチングを高得点にして、mismatch にも gap open にも寛容な設定。マッピングされる数は増えるが、どのようにマップされたか igv でみてみると、こんな感じ。
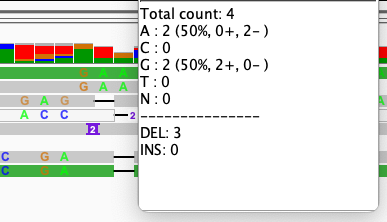
ある塩基には、7 つのリードがマップされている。うち A が 2 つ、G が 2 つ、gap (DEL) が 3 つ。これでは話にならないほど正確性が低い。
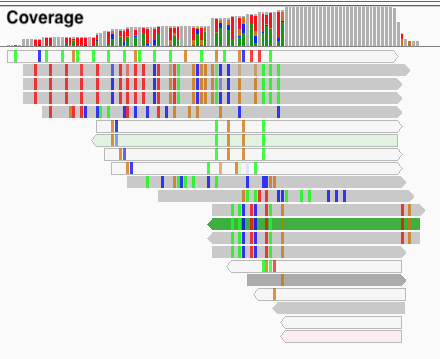
a = 1, b = 1, q = 5 の結果。上記と同じマッピングだが、領域は違う。全体を見渡して、適当と思われる部分のスクリーンショットを撮っている。
Coverage で灰色の領域があって良さそうに見えるが、実はこの領域はリピート。同じリピートをもつリードがここにマップされ、左側の方ではミスマッチがたくさんある。この場合、上の 3 つは同じ領域だろうが、それ以外はたぶん違うだろう。
ということで、この設定でも緩すぎると思われる。
広告
References
- BWA, NGS Surfer's Wiki. Link: Last access 2022/04/22.
- BWA, Bioinformatics. Link: Last access 2022/04/22.
- RUNNING BWA COMMANDS. Link: Last access 2022/04/26.
コメント欄
サーバー移転のため、コメント欄は一時閉鎖中です。サイドバーから「管理人への質問」へどうぞ。
