マッピングの結果を可視化する igv:
インストール、使い方など
- 概要: igv とは
- igv の結果の見方
広告
概要: igv とは
Integrative Genomics Viewer (igv) は、bowtie2 や bwa などによるマッピングの結果を可視化するめによく使われるソフトである。

igv のインストール
2022 年 5 月現在、このページ からダウンロード可能。
Linux では、ダウンロードされる zip ファイルを解凍し、含まれている igv.sh をターミナルから実行すると立ち上がる。
広告
「あとがき」で当サイトを参考にしたと書いてくれているラノベです。Kindle Unlimited で読めました。ストーリーと文章が良く、面白かったです。
igv の使い方
このページ にわかりやすく書かれている。マッピングの結果の bam ファイルを以下のようにソートし、index を作成する必要がある。samtools のページ も参照のこと。
samtools index output.bam
samtools index の際は、アウトプットに書き出す必要はない。上記を実行すると、output.bai というインデックスファイルが生成される。
ソートおよびインデックスの作成が終わったら、igv を開く。
Genomes – Load Genome file から、マッピングに使った reference genome のファイルを読み込む。gz 圧縮ファイルは使えず、解凍して fasta にしておく必要がある。
Reference genome を読み込んだら、File – Load from File で index 作成済みの bam ファイルを読み込む。
Reference genome が複数の配列に分かれている場合は、ウィンドウの左上から配列を選ぶことができる。染色体 Chromosome または contig に分かれていることが多いだろう。
igv の結果の見方
一番上に Coverage という行があり、ここでマッピングの概要を掴むことができる。Coverage では、
- Reference と同じ塩基は灰色
- Reference と異なる塩基は、それぞれの色
で表され、さらにそれぞれの色が割合の積み上げ棒グラフで示される。
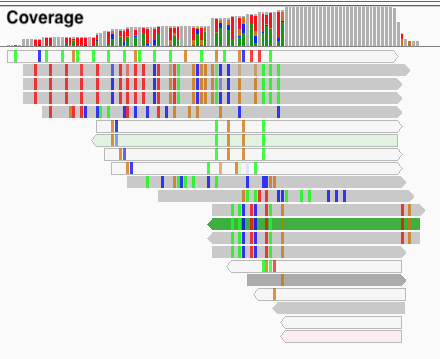
この図では、右の方に完全一致 (灰色) の領域があり、左の方でリードと Reference の配列にかなり違いがある。BWA でかなり条件を緩くしてマッピングした結果である。
広告
References
- igvtoolsはすぐれものーリードをカウントして各塩基の塩基組成のファイルを作る.Link: Last access 2022/05/17.
