R を使った生存曲線の描画: Kaplan-Meier
UB3/statistics/survival/survival_r
このページの最終更新日: 2025/11/23広告
R plot()関数を使った生存曲線の作成
このページ によると、R で生存時間分析に適したデータセットは以下の 3 つである。
- MASS パッケージに含まれている gehan
- survival パッケージに含まれている leukemia
- survival パッケージに含まれている veteran
ここでは、veteran パッケージを使った例を示しておく。これは肺がん治療のデータを示したデータセットで、以下のような項目を含んでいる (2)。本当は 137 行あるが、1 から 10 行目のみ記載する。
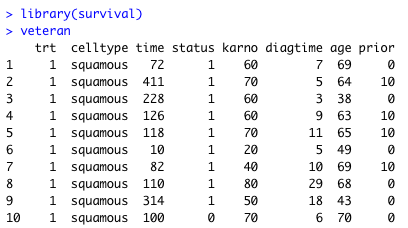
- trt: 治療の種類。1 が standard (1-69 行)、2 が test (70-137 行)。
- celltype: がん細胞の種類。
- time: 生存時間。
- status: 1 が打ち切りなし、0 が打ち切り。
- karno: Karnofsky performance score
- diagtime: 診断されてから clinical trial までの時間
- age: 年齢
- prior: Clinical trial 前の治療の有無。0 が治療なし、10 が治療あり。
survfit() で作成し、plot() で描画する
trt, time, status のデータを使い、Kaplan-Meier 曲線を作る。Surv() は、データを生存時間であると認識させる。これを survfit() 関数に入れて、Kaplan-Meier 曲線を作成。描画には plot() を使える。
plot(test_fit)
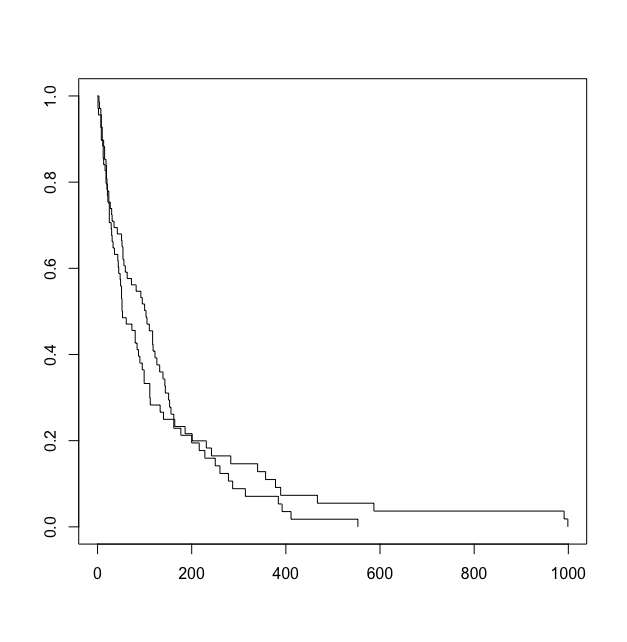
描画データは test_fit$surv というカラムに保存されているので、これを表示すれば最終的な生存率が何%であったかを知ることができる。
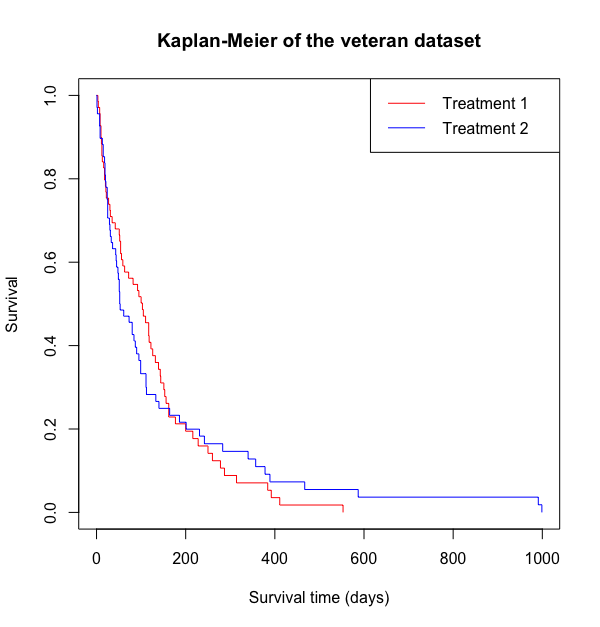
グラフの見た目は、plot() 関数のオプションを使って変更できる。詳細は plot 関数のページ を参照のこと。
labels <- c("Treatment 1", "Treatment 2")
plot(test_fit, main = "Kaplan-Meier of the veteran dataset", ylab = "Survival", xlab = "Survival time (days)", col = cols)
legend("topright", legend = labels, col = cols, lty = c(1, 1))
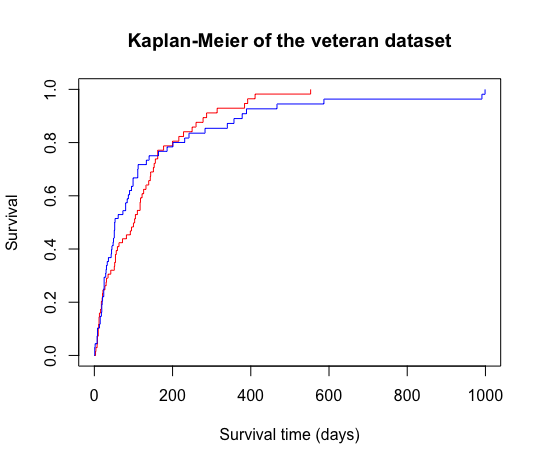
右上がりの Kaplan-Meier 曲線
plot に fun = "event" を追加すると、累積イベント割合、つまり右上がりの Kaplan-Meier 曲線になる。
labels <- c("Treatment 1", "Treatment 2")
plot(test_fit, main = "Kaplan-Meier of the veteran dataset", ylab = "Survival", xlab = "Survival time (days)", col = cols, fun = "event")
legend("topright", legend = labels, col = cols, lty = c(1, 1))
ggsurvplot() 関数を使った生存曲線の作成
ggplot() は、R できれいなグラフを作るための有名な パッケージ である。R base の plot ではなく、survminer というパッケージに含まれる ggsurvplot() 関数を使って生存曲線を図にすることもできる。
方法は同じで、test_fit を plot() でなく ggsurvplot() に渡す。
ggsurvplot(test_fit)
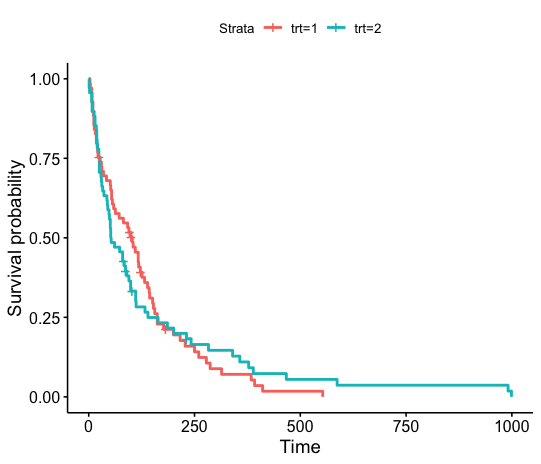
各種オプションは このページ に詳しい。
広告
References
佐藤弘樹、市川度. 2013. 生存時間解析がこれでわかる! 臨床統計まるごと図解.
|
生存時間解析 について平易に書いた数少ない解説書。 統計のなかでも、生存時間解析はそれだけで 1 冊の本になるほど複雑なわりに、ANOVAや t 検定などと違い使用頻度が低いため、とっつきにくい検定である。 この本では、とくに |
- R で生存時間分析を行う. Link: Last access 2018/09/20.
- Veterans' Administration Lung Cancer study. Link: Last access 2021/07/05.
- Survival Analysis. Link: Last access 2022/11/12.
- カプランマイヤー曲線の書き方 R と EZR の方法。Link: Last access 2024/04/24.
コメント欄
サーバー移転のため、コメント欄は一時閉鎖中です。サイドバーから「管理人への質問」へどうぞ。
