主座標分析 PCoA: 原理、方法など
UB3/statistics/pca/pcoa
このページの最終更新日: 2025/11/23- 概要: 主座標分析とは
- cmdscale() 関数による主座標分析
- 主座標分析の結果を平面上にプロットする
- Mardia fit measure による検証
- 腸内細菌叢の主座標分析 (β多様性)
- 負の値を含むデータ
広告
概要: 主座標分析とは
似た方法として 主成分分析 principal component analysis (PCA) がある。主成分分析はユークリッド距離を保ちながら次元を削減するが、主座標分析では他の距離や類似度が使える (1)。つまり、主成分分析は主座標分析の一種であると言える。
cmdscale() 関数による主座標分析
cmdscale() 関数の基礎
まず、使用する 組み込みデータセット は、ヨーロッパの都市間の距離データ eurodist。主座標分析の
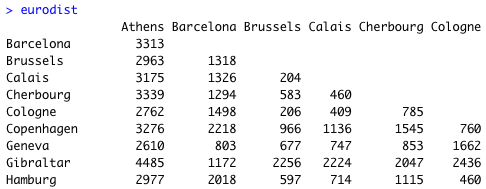
使う関数は cmdscale() で、これは距離行列を input とする。eurodist はもともと距離行列のデータセットなので、そのまま input にして良い。R の行列として読み込む。
result の中身はこのようになっており、それぞれの都市のもつ特徴が 1 と 2 の 2 つの数値で表されている。

k = 2 でこれを指定しているので、k = 3 にすると result はこのようになる。

eig = TRUE を指定した場合
cmdscale() 関数を使う際には、原則として以下のように
主座標分析の結果を平面上にプロットする
plot() 関数
このデータを平面上にプロットする。
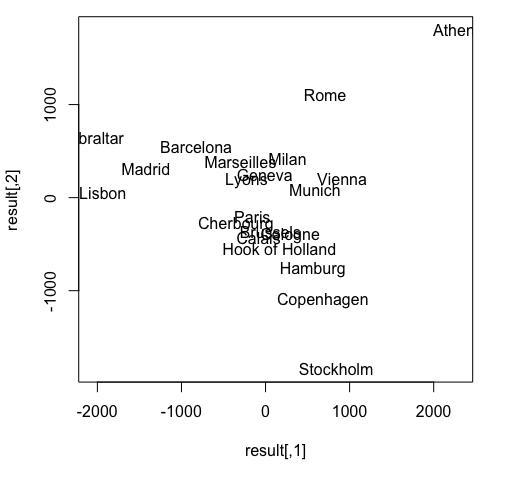
この 2 次元上の配置は、実際の都市の位置をよく反映している (1)。
ただし、この方法では以下の eig = TRUE を指定しているとエラーになる。文献 2 のように ordiplot を使う方が良いかもしれない。
ordiplot() 関数
vegan というライブラリに含まれる ordiplot() 関数でも描画できる。
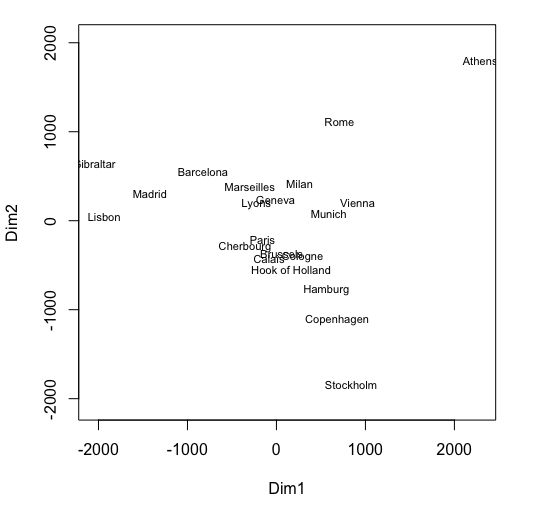
type は text, points, none のうちから選択。
PCA のように、この図に楕円を追加することもできる。ordiellipse という関数を使うようだ。いくつか参考リンクを置いておく。
ggplot() 関数
ggplot を使うのが一番きれいな気がする。
スクリプトなどの詳細は、まず このリンクでページの上の方へ行き、そこにある「β多様性について」をクリックしてください。
Mardia fit measure による検証
これも Ref. 1 から。eig = TRUE を指定することで、Mardia の基準 (Mardia fit measure) を計算できるようになる。
この eig は
主成分分析でよく使われる「寄与率」は、固有値を全項目の分散の合計で割った値であり、いわば eigen value を割合にしたものである。
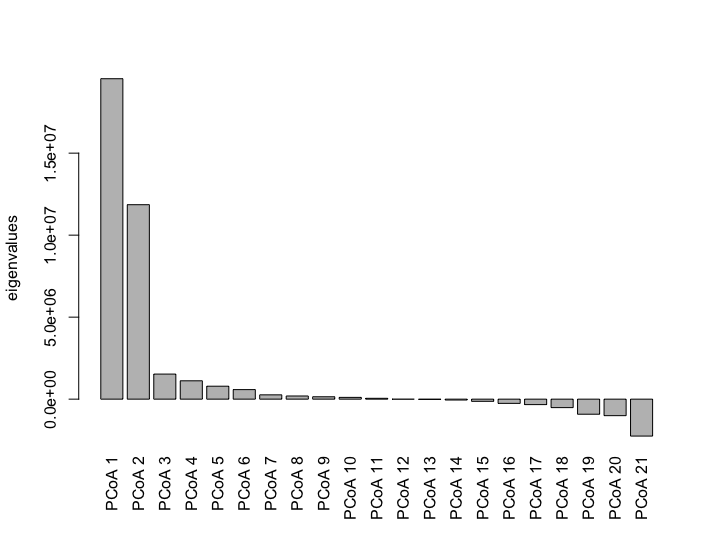
Mardia の基準 は、1 または 2 のどちらかが 0.8 以上であれば OK らしい (参考)。どちらも満たさない場合は、次元を増やす必要がある。そもそも情報を取捨選択しつつ次元を落としているのだがら、可能な場合と不可能な場合があることは合理的である。
腸内細菌叢の主座標分析 (β多様性)
生態学には β多様性という概念がある。これは、2 つのサンプルや地点間での生物多様性を示す指標で、2 つのサンプルがどれくらい似ているか (または似ていないか) が数値として与えられる。
他にも α多様性および γ多様性があるので、基礎的な内容については以下のサイト内検索から関連ページを参照のこと。

β多様性は距離であるので、β多様性の解析には主座標分析が用いられることが多い (PERMANOVA など、他の解析手法もある)。文献 3 に、腸内細菌叢の主座標分析について以下のように書かれている。要点をまとめて記しておく。
- 主座標分析は、腸内細菌叢を一つの生態系と見做し、生態系間の多様性の違いである
β多様性を視覚化する 方法である (参考: R を用いた β多様性の計算)。 - 主成分分析は、主座標分析の一種で、サンプル間のユークリッド距離を用いてクラスタリングする手法を指す。
- UniFrac 距離は、細菌の phylogenetic distance を加味した距離計算方法なので、これは PCA でなく PCoA にあたる。
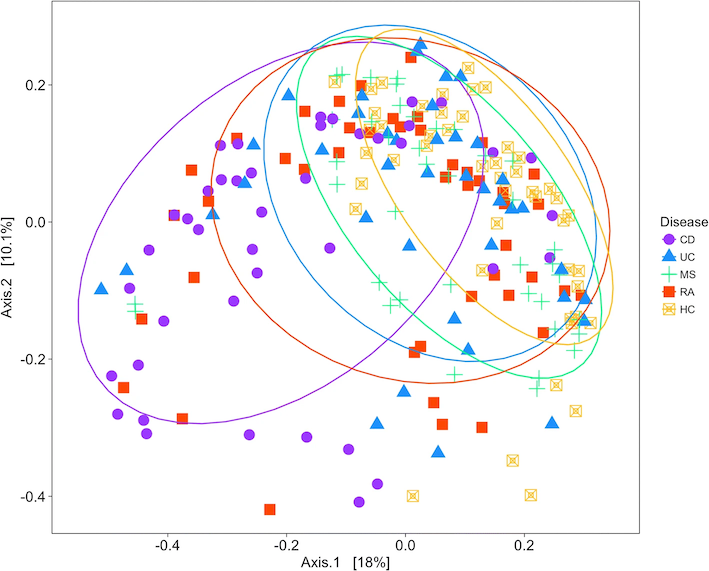
腸内細菌叢の主座標分析結果は、一般に次のような図で表される (4)。それぞれの点が個人を示し、腸内細菌叢が近い人が近くにプロットされている。楕円は 80% 信頼区間 を示す。
負の値を含むデータ
vegdist() 関数で距離を計算していて、かつデータが負の値を含む場合、results may be meaningless because data have negative entries in method “horn” のようなエラーメッセージが表示される。
vegdist() は、基本的に生物群集の量を解析する生態学パッケージであり、距離計算を目的としたパッケージではない。生物の存在量は負の値をとらないので、「計算することはできるが、結果は意味ないかもしれないよ」というメッセージである。
マニュアルには以下の記述がある。
|
Most dissimilarity indices in vegdist are designed for community data, and they will give misleading values if there are negative data entries. The results may also be misleading or NA or NaN if there are empty sites. In principle, you cannot study species composition without species and you should remove empty sites from community data. |
ただし、この GitHub ページ によると、Gower, altGower, mahalanobis は負の値も扱えるようである。altGower では上記のエラーメッセージが表示されるが、これは vegdist のエラーのようだ。
広告
References
- 主座標分析について簡単に紹介するよ! Link: Last access 2022/07/09.
- PCoA & NMDS, distance-based unconstrained ordination.Link: Last access 2023/01/01.
井上 2019a. 腸内細菌叢解析のいろは. Jpn J Lactic Acid Bact 30, 27-30.Forbes et al., 2019a. A comparative study of the gut microbiota in immune-mediated inflammatory diseases—does a common dysbiosis exist? Microbiome 6, 221.
コメント欄
サーバー移転のため、コメント欄は一時閉鎖中です。サイドバーから「管理人への質問」へどうぞ。