次世代シークエンシング: イルミナの原理と paired-end
UB3/informatics/bioinformatics/ngs_paired_end
このページの最終更新日: 2025/11/23- イルミナシークエンシングの原理
- Paired-end とは
広告
イルミナシークエンシングの原理
このページでは、イルミナシークエンシングの原理と paired-end についてまとめる。いずれ、さらにページを分割するかもしれない。
イルミナシークエンシングでは、まず DNA を断片化しタグをつける (図; ref 7)。
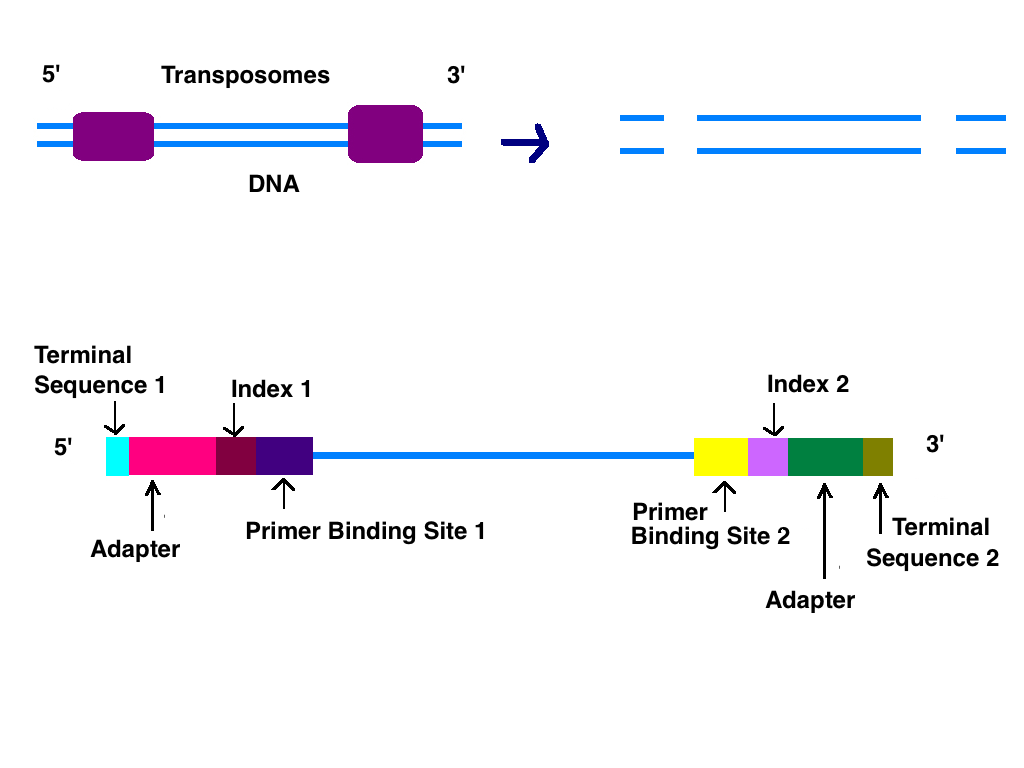
Illumina のフローセルには、タグ配列と相補的なオリゴヌクレオチド (図; ref 8) が並んでおり...
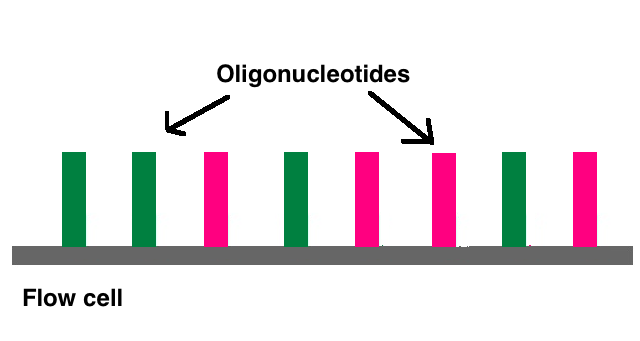
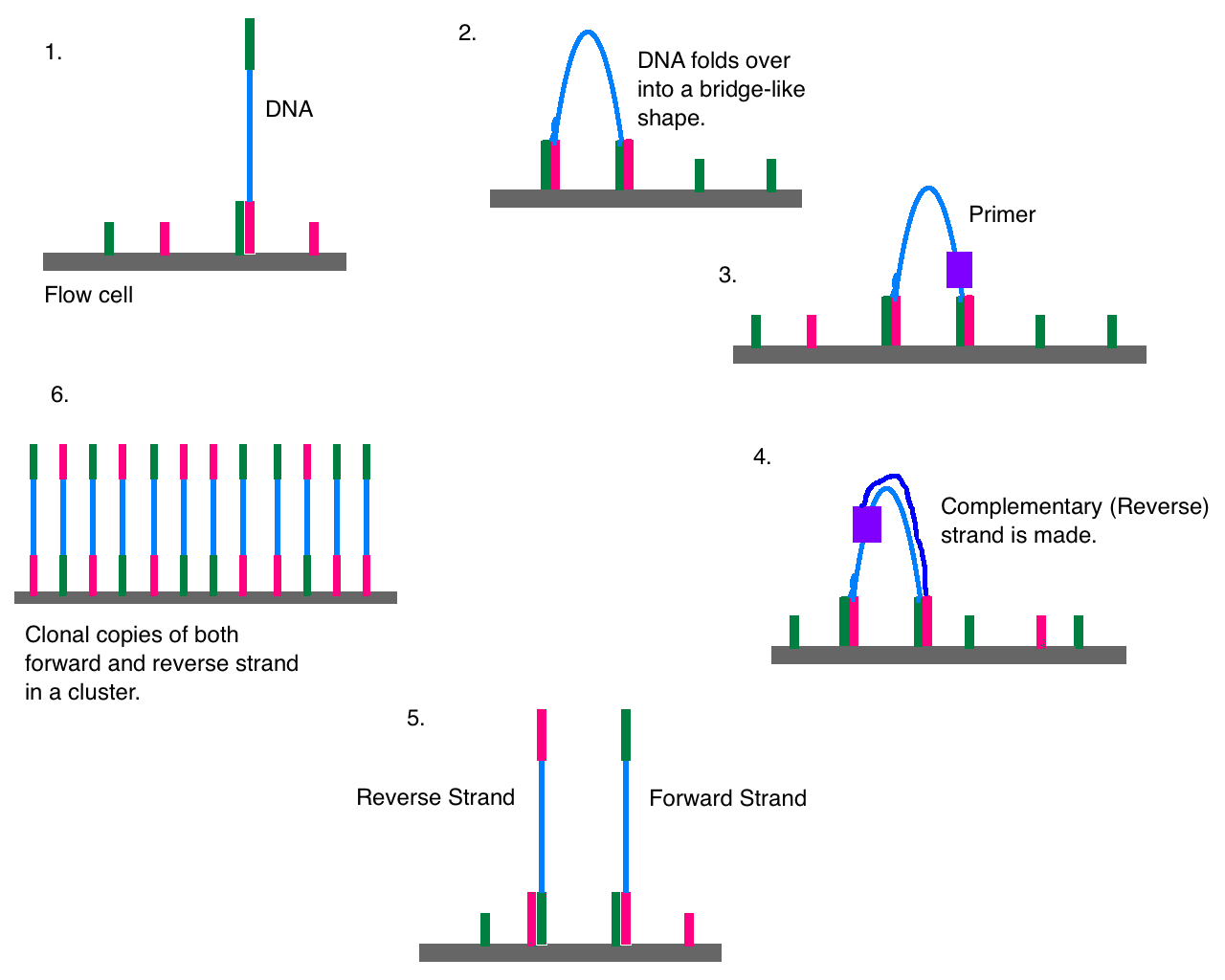
DNA と図 (ref 9) の 1 および 2 のようにハイブリする。ここにプライマーを加え、PCR をする。
PCR で使われる dNTP は、蛍光ラベルされている。配列を読む過程を箇条書きにしてみる。
- 蛍光ラベルした 4 種の dNTP のうち、1 種類を加える。
- たとえば dATP を加えると、もとの配列が T であるスポットにのみ dATP の伸長反応が起こる (図, ref 10)。
- 反応後、未反応試薬を除去し、蛍光を観察する。TT と 2 塩基連続していれば、蛍光強度も 2 倍になる。
- 蛍光を除去し、次のサイクルへ進む。
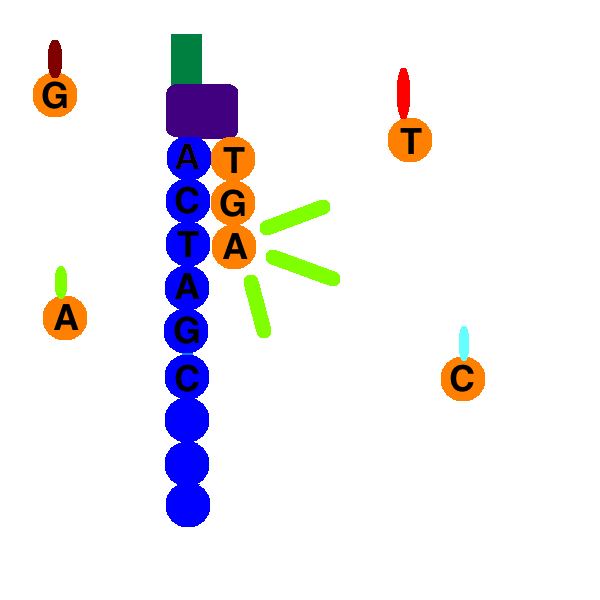
次世代シークエンシングのポイントは、以上の反応を 4 千万個以上 (11) 並列して行えることである。これによって、短時間に大量の塩基配列を得ることが可能になる。
広告
「あとがき」で当サイトを参考にしたと書いてくれているラノベです。Kindle Unlimited で読めました。ストーリーと文章が良く、面白かったです。
Paired-end とは
イルミナシークエンシングの原理の最初の図では、一つの DNA 断片の両側にタグをつけている。このような処理を行うことで、DNA を両側から読むことが可能になる。このように、両側から読む方法を paired-end sequencing という。これに対し、配列を片側からのみ読む方法は single-read sequencing と呼ばれる。
また、single-read sequencing によって決定された配列は single read, paired-end sequencing によって決定された配列は paired-end read である。
Paired-end read には、それぞれの配列自体がもつ情報に加えて、
- ライブラリ作製の際に DNA の長さを大体揃えるので、2 つの配列間の距離は大体どれくらいかわかる。文献 3 では「配列は 35 bp、両者の距離が 500 bp 程度」という例で説明されている。多くのイルミナ系シークエンスでは、これぐらいのオーダーと考えて良い。
- 上記の例で、2 つの配列が 1000 bp 離れて mapping された場合、その間に欠失があったことが予想される。
- この距離情報は、リピート配列に map する場合にも重要。
広告
References
ページ分割のため本文中に引用されていないものがありますが、番号と本文は対応しています。
- Amazon link:
Pevsner 2016. Bioinformatics and Functional Genomics.
- Amazon link:
清水、坊農 2019. 次世代シークエンサーDRY解析教本: 使っているのは第1版ですが、改訂第2版を紹介しています。
- Paired-End Read って何ですか? Link: Last access 2020/12/12.
- ページ編集に伴い削除
- ページ編集に伴い削除
- ページ編集に伴い削除
- By DMLapato - Own work, CC BY-SA 4.0, Link
- By DMLapato - Own work, CC BY-SA 4.0, Link
- By DMLapato - Own work, CC BY-SA 4.0, Link
- By DMLapato - Own work, CC BY-SA 4.0, Link
- イルミナシーケンサーの原理, イルミナ株式会社. Pdf file.
コメント欄
サーバー移転のため、コメント欄は一時閉鎖中です。サイドバーから「管理人への質問」へどうぞ。
