R で同義置換率を計算する: kaks function
UB3/informatics/r/synonymous
このページの最終更新日: 2025/11/23- 概要: 同義置換・非同義置換とは
- R を使った計算の実際
- 配列の準備
- アラインメント
- R への読み込みと kaks
広告
概要: 同義置換・非同義置換とは
mRNA は、以下のコドン表 (Public domain) にまとめたような規則に従ってアミノ酸に翻訳される。とくに 3 番目の塩基に多くみられるように、
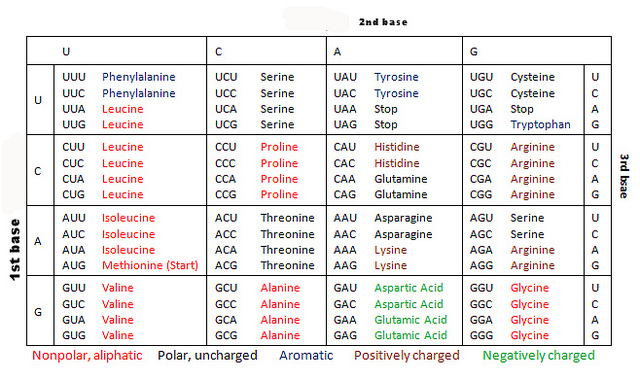
塩基の置換のうち、コードされるアミノ酸が変わらないような置換を
このページには、R を使った実際の計算方法を示す。同義置換・非同義置換の基礎的な項目については、以下のサイト内検索結果を参照のこと。

seqinr パッケージのインストール
R がインストールされていなければ、R の概要 のページに従いインストールする。同義置換・非同義置換率は kaks function で計算できるが、これはデフォルトのパッケージに含まれていないため、以下のように seqinr というパッケージをインストールする必要がある。
パッケージ名の指定には " " が必要である。シングルクオーテーション ' ' でも OK なようである。パッケージのインストール のページも参照のこと。
Seurat
広告
「あとがき」で当サイトを参考にしたと書いてくれているラノベです。Kindle Unlimited で読めました。ストーリーと文章が良く、面白かったです。
R を使った計算の実際
配列の準備
同義置換・非同義置換の計算には、
まず、PubMed から適当な配列をダウンロードし、BLAST で orthologues を何個か取ってくる。これを Clustal Omega でアラインメントし、phylip 形式で保存する。
今回は、牛乳の主要タンパク質であるカゼイン casein を例にしてみる。
アラインメント
このモルモットカゼインを BLASTN し、orthologue を適当に 3 つ選んで、計 4 個を FASTA で保存。これを Clustal Omega で output を phylip にしてアラインメント。Download Alignment File から phylip ファイルをダウンロードする。名前は test.phylip とする。

R への読み込みと kaks
まず、R の read.alighment 関数でこの phylip ファイルを読み込み、変数に格納する。setwd() ファイルのある場所を作業ディレクトリとし、
のようにする。エラーが出なければ成功である。phylip2.res として変数の中身を覗いてみると、以下のようになっているはずである。
あとは、kaks function でこの phylip2.res を解析するのみ。
コマンドはこれで合っているが、そのままやると sequence lengths are not a multiple of 3 というエラーになった。ある意味これは当然で、塩基配列が 3 の倍数でないと計算不能である。配列は注意深くトリムする必要がある。
うまく kaks が動けば、同義置換・非同義置換率は総当たりで計算される。
広告
References
コメント欄
サーバー移転のため、コメント欄は一時閉鎖中です。サイドバーから「管理人への質問」へどうぞ。