R を使った one-way ANOVA および post-hoc test
UB3/informatics/r/r_anova
- R を使った one-way ANOVA (パラメトリック)
- R を使った post-hoc test (パラメトリック)
- R を使った Kruskal-Wallis (ノンパラメトリック)
- R を使った post-hoc test (ノンパラメトリック)
- Steel-Dwass test
広告
R を使った one-way ANOVA
このページでは、R を使った一元配置分散分析 one-way ANOVA の方法を解説する。少なくとも R version 3.5.1 から 4.0 までは使える方法である。ANOVA および post-hoc テストの原理については、以下のページを参照のこと。
R では、ANOVA に aov および anova 関数を用いる。まずあるオブジェクト (仮に D とする) に aov の結果を格納し、それをさらに anova で解析するという段階を踏む。D を別の関数 TukeyHSD などに入れると post-hoc テストを引き続き行うことができる。
ベクターを input にする ANOVA
データ形式は、数値とグループの 2 つのベクターを使うのが簡単である。つまり、数値のベクター (1, 1, 1, 2, 2, 2, 3, 3, 3) とグループのベクター (A, A, A, B, B, B, C, C, C) のような形式である。
test_value = c(1, 2, 1, 7, 8, 6, 34, 64, 55)
D = aov(test_values ~ test_group) # aovで解析
anova(D) # Dをさらに anova で解析
最初の 2 行で、グループおよび数値のベクターを作っている。A, B, C の 3 つのグループがあり、A は 1, 2, 1 という数値、B と C にはそれぞれ 7, 8, 6 および 34, 64, 55 というデータである。したがって扱うデータは 9 つであり、平均値 は C > B > A の順に大きい。
数値を test_values というベクターに格納し、グループ分けを test_groups というベクターに格納している。なお、test_groups を作るとき A, B, C は文字列なので、" " で囲む必要がある。
これを aov で解析し、結果を D という変数に保存する。test_values ~ test_group の部分は、最初が数値のベクター、次がグループのベクターという順にする。
下のスクリーンショットのように、D という変数を使わずに、直接 aov を anova に入れても OK。
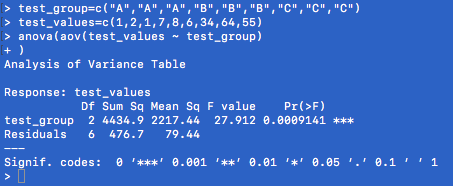
結果として表示されるのは、自由度 Df、偏差平方和、平均平方、F および P 値であり、P 値が有意水準以下であるかどうかを判断する。
統計表現のページ にあるように、F 値、P 値および自由度を論文に記載するとよい。
データフレームを input にする ANOVA
values と group が、df というデータフレームの列である場合には、以下の書き方で aov をすることができる。R の一般的な書き方である。それ以降は同じ。
anova(D)
広告
「あとがき」で当サイトを参考にしたと書いてくれているラノベです。Kindle Unlimited で読めました。ストーリーと文章が良く、面白かったです。
R を使った post-hoc test
Tukey HSD test
Tukey HSD は R にもともと含まれているようで、単に aov の結果を TukeyHSD という関数に入れるだけでよい。以下の例では、まず aov を AResults という変数に格納している。
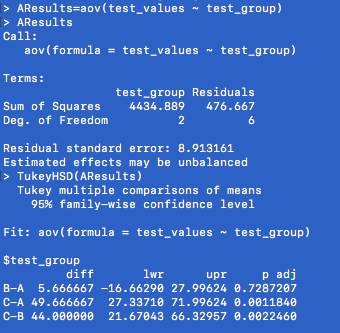
なお、Tukey 法は各群の n が揃っているときのみ使えるが、Tukey-Kramer 法は n が同じでも違っていても使うことができる。R の TukeyHSD 関数は Tukey-Kramer なので、n は心配せずにこれを使えばよい。
Dunnett's test
Dunnett's test は、対照群とその他の群を比較する際に使われる post-hoc test である。まず、この関数が含まれているパッケージをインストールする必要がある。
2021 年 12 月の解析では、install.packages("multcomp") のみで大丈夫だった。囲み部分は古い情報なので、multcomp がインストールできれば飛ばして構わない。
|
2018 年 12 月の実行記録、R version 3.5.1, Mac OS 10.14. このページ から multcomp というパッケージを .tar file としてダウンロードし、ダウンロードフォルダで R CMD INSTALL ”.tarファイル名" でインストール。さらに library(multicomp) で読み込もうとすると、package ‘mvtnorm’ required by ‘multcomp’ could not be found というエラーになった。mvtnorm というパッケージが必要なようだ。 そこで、ここ から mvnorm をダウンロード、同様の方法でインストールするが、何かが not found というエラーに。 別の方法として、このページ にあるように として URL 指定すると、なぜか library(multicomp) もエラーが出ずに成功する。この際、次のようなメッセージが出る。
multcomp_1.4-8, TH.data_1.0-9, MASS_7.3-50, survival_2.42-3, mvtnorm_1.0-8 がインストールされた模様。コンピューターによっては、 として mvtnorm パッケージを別にインストールしないと library(multcomp) が成功しなかった。 |
multicomp がインストールされたら、以下のように解析を実行。y は従属変数 (つまり数値)、x は独立変数 (つまりグループ) である。
summary(glht(aov(y ~ x), linfct=mcp(fx="Dunnett")))
# x, y がデータフレーム df の列のとき
summary(glht(aov(y ~ x, data = df), linfct=mcp(df$x="Dunnett")))
mcp(df$x="Dunnett") の部分では、df$x の部分は説明変数になる。ここの名前が列名と違っていると、「その列がモデルにみつからない」という以下のようなエラーになる。
Error in mcp2matrix(model, linfct = linfct) : Variable(s) ‘fx’ have been specified in ‘linfct’ but cannot be found in ‘model’!
また、ここで x はカテゴリー変数になる。データが数値で、数値として認識されていると以下のようなエラーになる。
Error in mcp2matrix(model, linfct = linfct) : Variable(s) ‘x’ of class ‘numeric’ is/are not contained as a factor in ‘model’.
この場合は、x をカテゴリーに変えてやらなければならない。df$x <- as.category(df$x) とする。
というか、そもそも x が数値ならば、それは x と y ともに数値データとなるので、ANOVA よりもむしろ 回帰分析 を考えるべきデータである。
Duncan test
ページの上に リンク がある「post-hoc test の選び方」のページで述べているように、Duncan test (Duncan's new multiple range test) は、第一種の過誤 (type I error) を適切にコントロールしていないので、有意差が出やすく、推奨されない。
しかし、方法を一応ここに示しておく。この問題点は、Duncan test の RDocumentation でも触れられている。
関数は duncan.test()、agricolae パッケージが入っていなければ、インストールする必要がある。Documentation にしたがって、sweetpotato というデータセットを使う。
sweetpotato は、おそらく agricolae パッケージに含まれるデータセットで、ウイルスの種類とスイートポテトの収量が含まれるシンプルなデータセットである。

これに Duncan test を実行する場合の syntax は以下の通り。意外とややこしい。
data(sweetpotato)
model <- aov(yield ~ virus, data = sweetpotato)
df <- df.residual(model)
MSerror <- deviance(model)/df
out <- with(sweetpotato, duncan.test(yield, virus, df, MSerror, group=TRUE))
plot(out,horiz=TRUE,las=1)
print(out$groups)
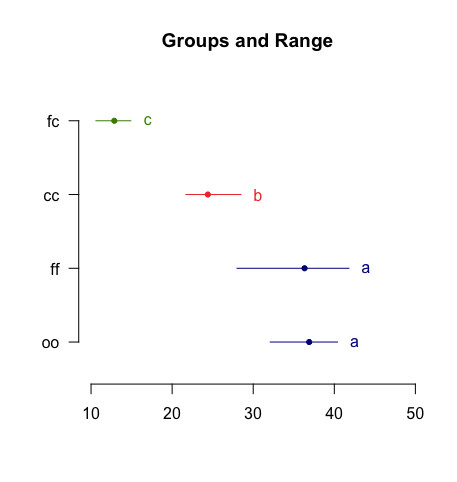
実行すると、以下のように結果とプロットが出力される。有意差が論文のように a, b... で示されるのはちょっといい感じである。

R を使った Kruskal-Wallis テスト (ノンパラメトリック)
更新予定。
R を使った post-hoc test (ノンパラメトリック)
このページ を参考に実行した。いずれ別ページでまとめる。
広告
References
コメント欄
サーバー移転のため、コメント欄は一時閉鎖中です。サイドバーから「管理人への質問」へどうぞ。
