R: ComplexHeatmap でヒートマップに列アノテーションを追加する
UB3/informatics/r/heatmap_annotation_col
このページの最終更新日: 2025/12/15広告
データ準備
ComplexHeatmap 関数で、ヒートマップの上または下にアノテーションを追加する方法をまとめる。heatmap の列に対して情報を追加することになるので、
ヒートマップに行アノテーションを追加する 方法とはけっこう違っている。
まずは、このページ を参考にデータセットの作成。少し改変している。
TestData1 は、Gene 1 から Gene 5 の発現量が格納された数値データ。Category は 0 または 1 の変数。
TestData2 は、それぞれの値に A から Z までのアルファベットを割り振っている。Gene 1 にはときどき NA が入る。
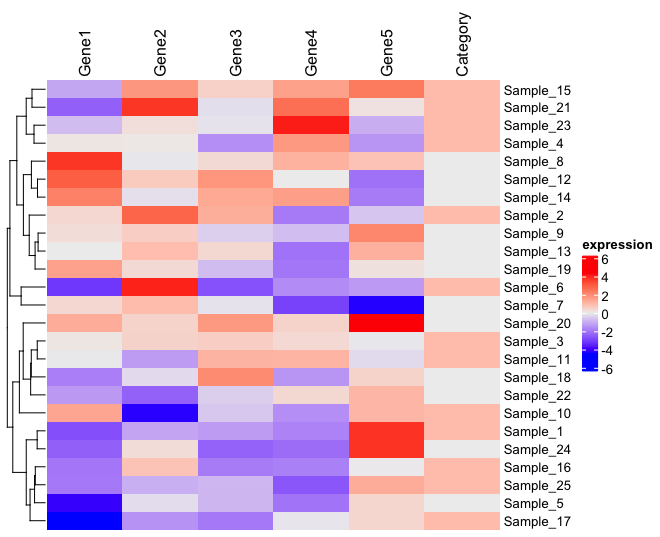
TestData1 のヒートマップは、以下のようになる。
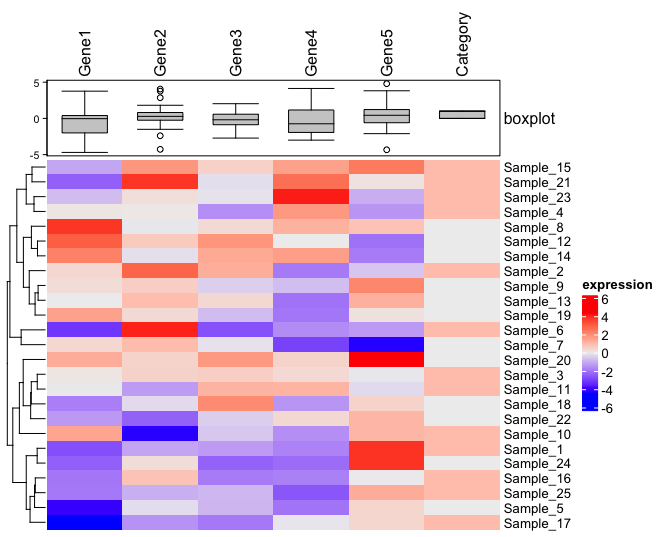
ボックスプロットを追加する
基本的には、列アノテーションは HeatmapAnnotation という関数を使って作図し、それを変数に格納、Heatmap 関数で参照するという形をとる。
Heatmap(TestData1, cluster_columns = FALSE, cluster_rows = TRUE, row_names_gp = gpar(fontsize=10), column_names_side = "top", name = "expression", top_annotation = TopAnotation)
このままだとカテゴリーまでボックスプロットに入ってしまっているので、それは行アノテーションとして別にする。
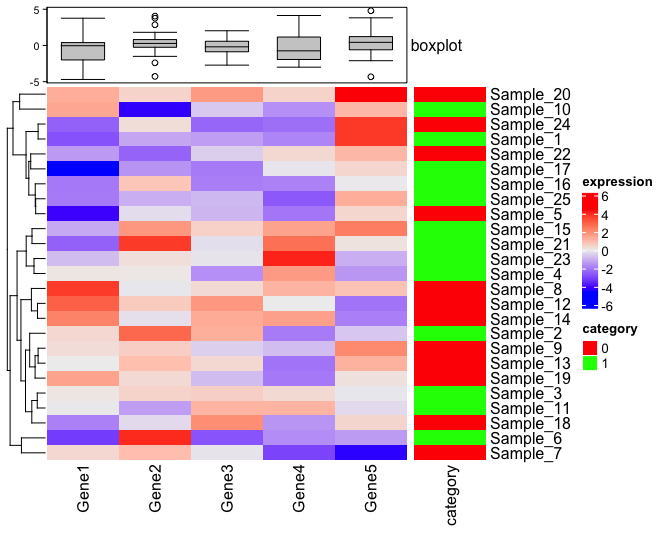
HeatmapAnnotation 関数にどのようなオプションがあるかが重要になる。
広告
「あとがき」で当サイトを参考にしたと書いてくれているラノベです。Kindle Unlimited で読めました。ストーリーと文章が良く、面白かったです。
カテゴリーのヒートマップを追加
1 個追加
次に、Gene 1 と Gene 2 が alpha、Gene 3 から 5 が beta というグループになっているとして、その情報を加えてみよう。
HeatmapAnnotation の部分が少しわかりにくいが、このようなシンプルなベクターデータのときは、このようにただ追加すれば良さそうである。
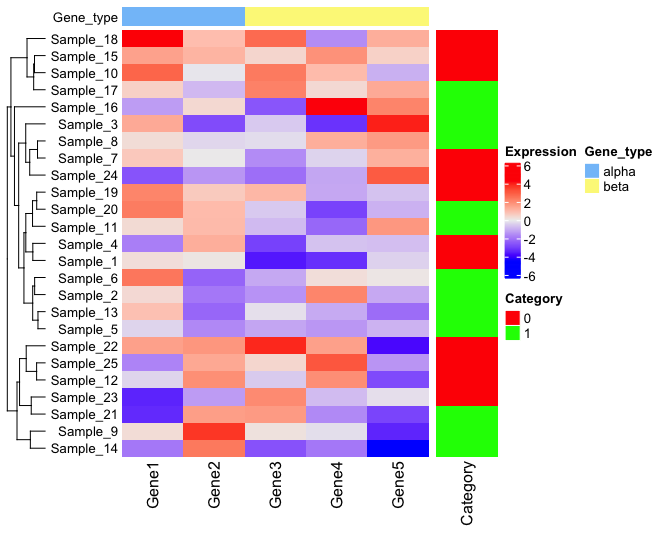
2 個追加
もう一つ別のカテゴリーを追加したい場合。上記の alpha と beta に加えて、Gene 1 か inducible, Gene 2 - 5 が constitutive というカテゴリーも追加してみる。
単に HeatmapAnnotation で 2 つ指定すれば良いようだ。
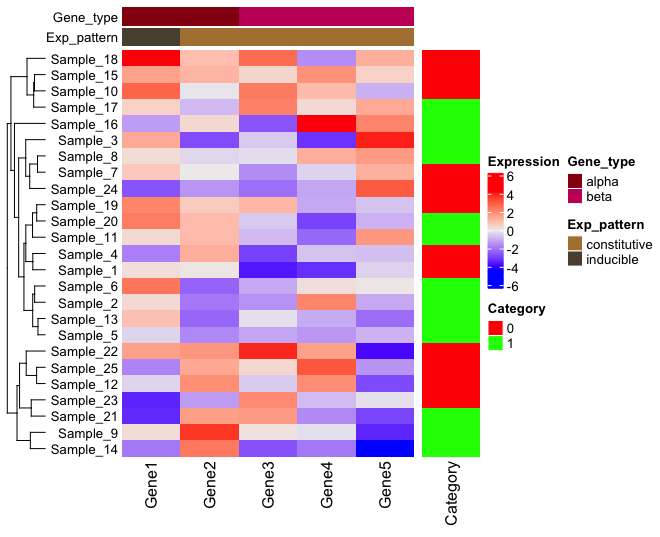
列のクラスタリングを入れると
width1 で cluster_columns を TRUE にして、列をクラスタリングすると、このように Gene 1 が 4, 5 とクラスタリングされ、中央に移動する。このとき、もちろん Annotation も一緒に動く。
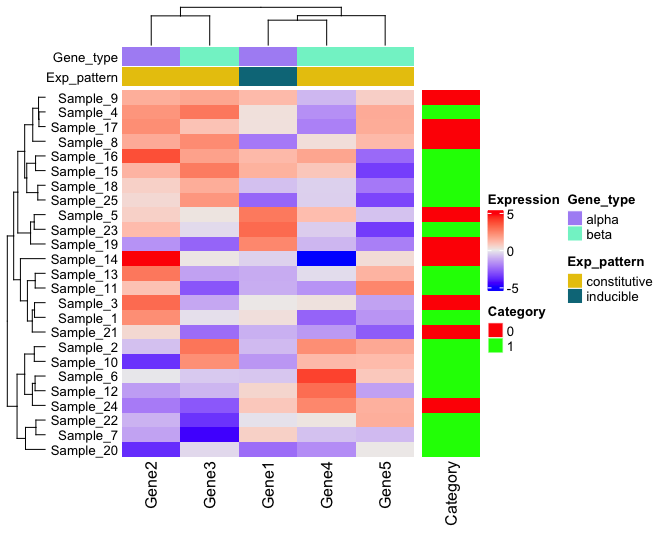
しかしながら、Gene_type と Exp_pattern で参照している gene_category と gene_category2 は単なるベクターであり、Gene 1, Gene 2... に紐づけられた情報はもっていない。
どうやってアノテーション情報と Gene 1, Gene 2... を関連づけているのか、気になったので少し調べてみた。
alpha, beta の分類を少し変えて、Gene 1, 2, 4 が alpha, 3, 5 が beta になるようにする。
すると、確かに 1, 2, 4 が alpha になったヒートマップができる。
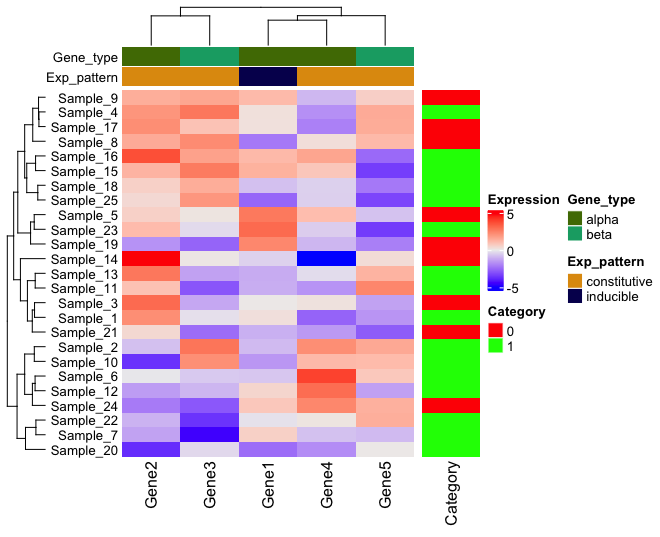
HeatmapAnnotation 関数の中で、gene_category を sort してみる。
この場合、Gene 1, 2, 3 が alpha, 4, 5 が beta となる。これを解釈すると、おそらく以下のような手順を踏んでいると思われる。
- HeatmapAnnotation を実行する時点で、もとのベクター (gene_category) が記録される。
- クラスタリングの前に、その順番でヒートマップのデータと関連づけられる。
- その後、クラスタリングを行えば Annotation とデータは一緒に動く。
したがって、Annotation 用のベクターと、heatmap 用の matrix の順番は一致していなければいけない。データフレームを読み込んで、順番がバラバラでも名前でマッチさせてくれるような期待をもつかもしれないが、そうでないことに注意。
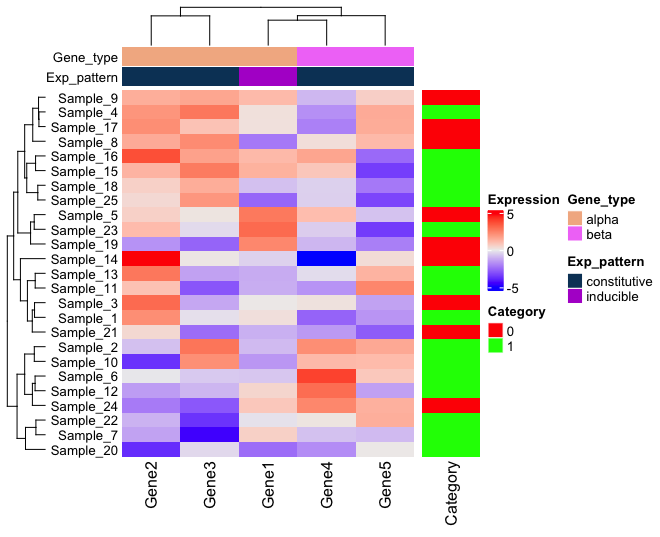
ただし、HeatmapAnnotation のところは df でデータフレームも読み込める。通常は 1 次元のデータフレームで、ベクターとほぼ同じ扱いになるっぽいが、ここに 2 次元以上のデータフレームを読み込んだらどう扱われるのか、まだちょっとわからない。
annotations should be vector/data frame (only `df`)/matrix/functions. というエラーが出たことがあるので、少なくともデータフレームを使う場合、名前は df でなければいけないようだ。
アノテーションの色を指定する
アノテーションが上の例のように alpha, beta などのカテゴリーの場合は、col のなかに list を作って一つずつ色を指定する。連続変数の場合は別の指定方法がある。
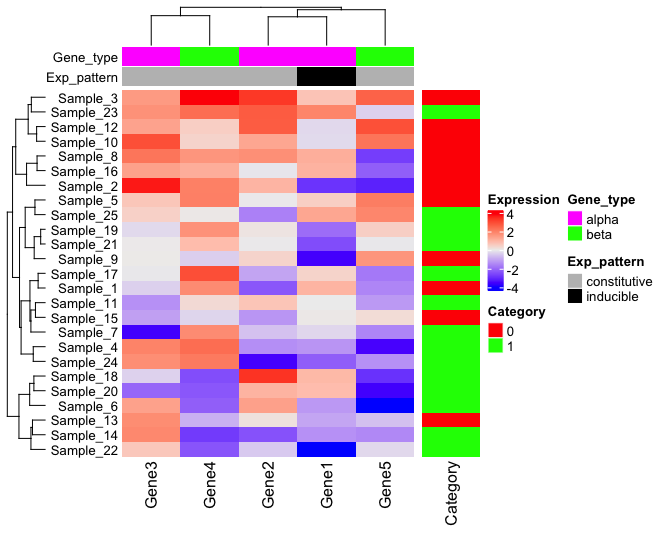
アノテーションの legend を変更する
アノテーションの legend は、デフォルトではアルファベット順になる。HeatmapAnnotation 内で、annotation_legend_param を指定することで変更できる。
at で順番を変更することで、legend の表示順も変えることができる。Gene 1 が inducible で、黒で表されているが、これが i と表示されて上になっていることをチェック。ヒートマップでは、依然として Gene 1 が黒になっているので、これは legend だけの変更である。
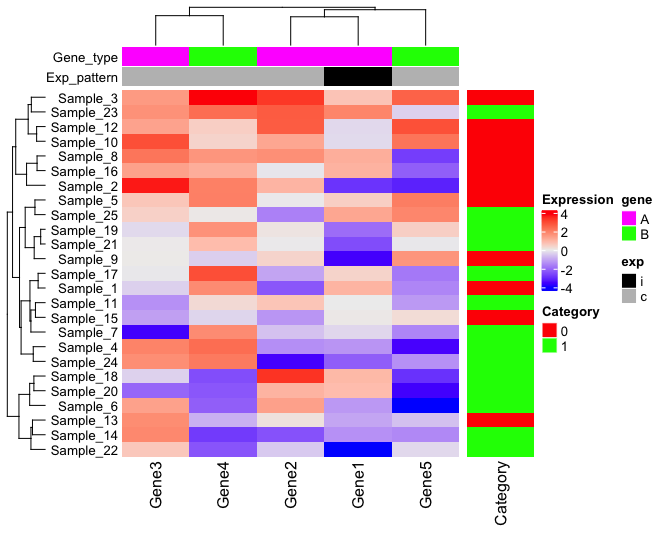
連続変数のヒートマップを追加する
Annotation = HeatmapAnnotation(Gene_type = sort(gene_category)... の部分で、連続変数のベクターを指定すれば、連続変数のヒートマップがアノテーションとして追加される。
広告
References
Script: ub_related/R_script/Practice_heatmap_2_v1.R
- R : matrixとdata.frameの違い. Link: Last access 2022/05/21.
コメント欄
サーバー移転のため、コメント欄は一時閉鎖中です。サイドバーから「管理人への質問」へどうぞ。
