R の dbrda() 関数
UB3/informatics/r/dbrda
このページの最終更新日: 2025/11/23広告
概要: dbrda() 関数とは
dbrda は、群衆組成の違いを環境変数で説明する関数である (3)。どうも重回帰分析のようである。
dbrda についてのほぼ唯一の日本語資料である文献 3 の内容をそのままなぞるだけだが、このページにまとめてみる。
組み込みデータセットを使っているのかと思ったが、このページ にあるサンプルデータを使っている。csv ファイルとしてダウンロードする。
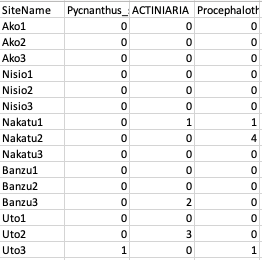
spcdat.csv は、個体数のデータである。縦にサンプル (採集地点、ヒトのマイクロバイオーム解析なら被験者になるだろう)、横に species がきて、数値は個体数 abundance である。15 地点、102 種を含む 15 x 103 のデータであるが、一部のみ例として載せておく。
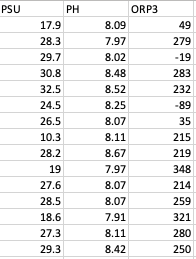
envdat.csv は、同じ 15 地点の環境変数のデータである。地点の情報は含まれていないが、この 15 行は spcdat.csv の 15 地点に対応している。11 種の環境変数があるため、15 行 x 11 列のデータとなっている。
これらをそれぞれ dat, edat として読み込み、文献 3 「Rによる群集組成の解析」にしたがって解析する。ここでは dbrda の使い方がメインなので、変数の分布チェックや変換などは飛ばす。
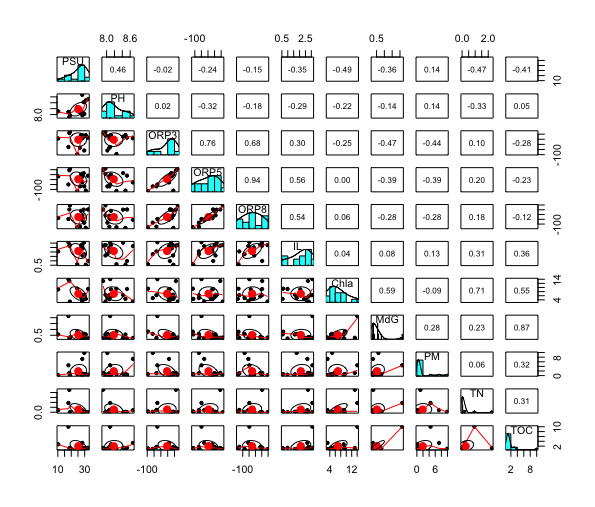
一応 psych でチェックすると、ちゃんと相関が出力される。
さらに文献 3 の通りに
とすると、Error in dfun(X, distance) : input data must be numeric というエラーメッセージ。SiteName という列が文字列データなので、これがいけないのかと思い dat2 を以下のように設定して再トライ。一応結果が出力された。
とりあえずこの関数を動かすためには、サンプル番号なしの metadata (環境要因の数値データ) と、サンプル番号を含む species data (種ごとの個体数データ) が必要であることがわかった。
文献 3 では、続いて anova.cca という関数を使っている。重回帰であるところの dbrda の有意性を調べているようである。RDocumentation によると、The function performs an ANOVA like permutation test と書かれているので、どうも permutation test という検定を行っているらしい。
Permutation test は、カタカナでパーミューテーション検定と呼ばれることもあるが、日本語に訳すなら「並べ替え検定」のようだ。このページ の説明がわかりやすい。
もともとは 2 群の検定であり、両群のデータ数は固定したまま、データをランダムに入れ替える。入れ替えた群について統計値を算出し、オリジナルの統計値と比較する。2 群に本当に差があるならば、データ入れ替え後には、オリジナルの群よりも有意になる確率が低いはずである。考え方としては、ランダムサンプリングによって新しい群を作る bootstrap 法 と似ている。anova.cca は、複数の群でこれを行うテストである。
さらに、ordistep() 関数を用いて変数選択を行っている。これで変数を減らすと、説明可能な部分は減るが、回帰自体は有意になることが示されている。この ordistep() には、ordiR2step() というバリエーションもあり、こちらでは adjusted R2 と P 値に基づいた forward model choice のみを行う。
これらの関数についての参考ページ。
- vegan::ordiR2step() doesn't find best-fit model
- ordiR2step tossing the last factor
- sel.fs という似た関数のページ
広告
「あとがき」で当サイトを参考にしたと書いてくれているラノベです。Kindle Unlimited で読めました。ストーリーと文章が良く、面白かったです。
どんなプロットが論文に載っているか
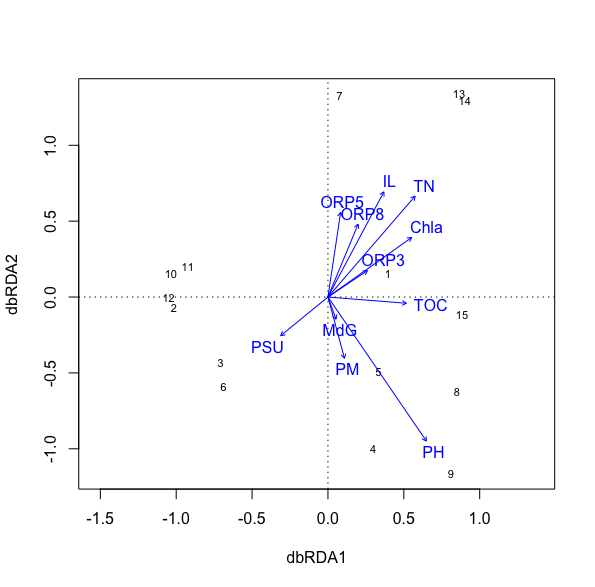
作図の参考にするため、論文から RDA 解析の図を拾ってみる。
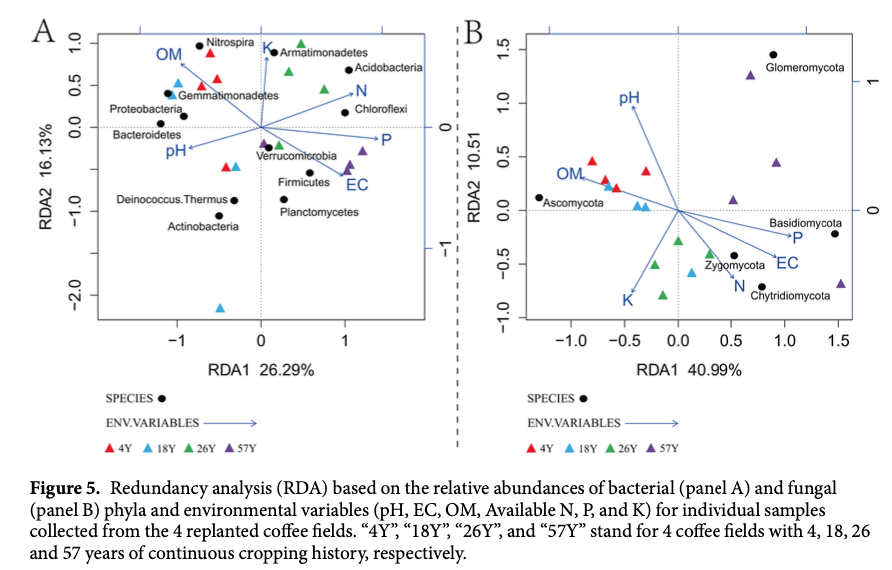
まずは Zhao et al. Sci Rep 8, 6116, 2018 より。A はバクテリア、B は真菌がそれぞれプロットされており、矢印には pH、organic matter (OM) などの環境要因がある。4Y, 18Y などはサンプル。論文に書かれている解釈は以下の通り。
- まず、含めた環境要因がバクテリアおよび真菌で total variation のそれぞれ 42.42% および 51.50% を説明した。これは軸の RDA の横にある数字の和である。
- バクテリアでは、4Y と 18Y が近く、26Y および 57Y とは RDA1 で分けられる。26Y および 57Y は RDA2 で分けられている。
- Proteobacteria, Nitrospira, Bacteroidetes は 4Y, 18Y サンプルに多く、soil OM と相関が強い。平面上で近くにあるということ。
- Acidobacteria, Firmicutes は、それぞれ N および EC と associate している。
- 真菌でも同じような解釈が書かれているが、これは省略。
dbRDA メモ
下記のエラー。原因はいろいろあるかもしれないが、まずチェックするのは「全てが 0 であるサンプルがない」こと (2)。全てが 0 の場合、distance が計算できないのでこのエラーになる。
広告
References
- 最近読んだ論文のメモ: 環境要因と群集構造の比較法 (CCA, RDAなど). Link: Last access 2023/08/07.
- Error missing value where TRUE/FALSE needed when running metaMDS in R. Link: Last access 2023/10/24.
- Rによる群集組成の解析. Link: Last access 2022/05/10.
コメント欄
サーバー移転のため、コメント欄は一時閉鎖中です。サイドバーから「管理人への質問」へどうぞ。
